Le cycle viral de SARS-CoV-2
Par Matthew Borok. Institut Mondor de Recherche Biomédicale – Université Paris-Créteil
La famille des coronavirus
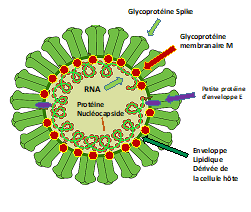
Les Coronavirus sont des virus dont le matériel génétique est un ARN simple brin linéaire à polarité positive. Ils doivent leur nom à leur apparence en forme de couronne (corona en latin) qui résulte de la présence de grandes glycoprotéines (protéines modifiées par des sucres) appelées spicules (Spike, S) enchâssées dans l’enveloppe du virus (Figure 2).
Trois coronavirus humains sont apparus depuis le début des années 2000 et ont provoqué des épidémies : SARS-CoV (2003), MERS-CoV (2012), et enfin SARS-CoV-2 (2019) (1). Cependant, seul le SARS-CoV-2 est en quelques mois devenu pandémique.
Liaison de SARS-CoV-2 et pénétration dans les cellules
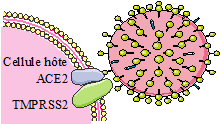
Le virus est un pathogène intracellulaire obligatoire, et doit pénétrer dans une cellule hôte pour pouvoir se multiplier (on parle de réplication). La première étape de ce processus est donc l’entrée du matériel viral dans le cytoplasme après avoir franchi la membrane cellulaire. L’étape d’entrée débute par l’attachement de la particule virale à la surface de la cellule. Celle-ci repose sur l’interaction entre les spicules à la surface de la particule virale (protéine S du SARS-CoV-2) et la glycoprotéine angiotensine-converting enzyme 2 (ACE2) qui agit en tant que récepteur d’entrée (Figure 3).
Après fixation à ACE2, la spicule virale (S) est coupée en deux parties par une protéase (enzyme qui coupe les protéines) de la cellule hôte. Cet évènement moléculaire est nécessaire pour exposer une partie de la séquence polypeptidique de S appelée « peptide de fusion » qui s’insère dans la membrane cellulaire. S’ensuit un rapprochement entre l’enveloppe du virus et la membrane cellulaire, toutes deux formées par une bicouche lipidique qui fusionneront donc (2). Parmi ces protéases, la molécule TMPRSS2 qui est présente à la surface de la cellule permet la fusion du virus avec la membrane plasmique de la cellule hôte. Le virus peut également entrer par « endocytose »: la fixation de Spike à ACE2 va induire une invagination de la membrane plasmique, englobant le virus qui rentre dans un « endosome » où une protéase, activée par l’acidité de ce compartiment, permettra de déclencher la fusion entre la membrane endosomale et la membrane virale. La fusion entre les membranes cellulaires et virales libère l’ARN viral dans le cytoplasme cellulaire où se met en place la réplication du virus (Figure 5).
La présence du récepteur viral est un déterminant majeur de la reconnaissance spécifique entre le virus et l’hôte (ou tropisme), c’est-à-dire la cellule, le tissu ou même l’espèce animale dans laquelle le virus peut se multiplier.
SARS-CoV-2 peut donc infecter les cellules humaines exprimant ACE2 : cellules du poumon, des artères, du cœur, des reins et de l’intestin.
Synthèse des composants du virus, assemblage et sortie de particules virales néo-synthétisées
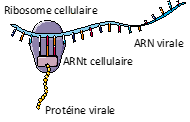
Une fois à l’intérieur de la cellule hôte, le virus va détourner les processus cellulaires (on parle aussi de machineries) de production de protéines (traduction) au profit de la synthèse de ses propres composants. L’ARN viral est traduit par les ribosomes (usines où l’ARN messager contenant l’information génétique est converti en protéine fonctionnelle). Ce processus met en jeu les ARN de transfert cellulaires (ARNt) qui mettent en correspondance un « codon » de trois nucléotides et un acide aminé donné (Figure 4).
Plus en détail :
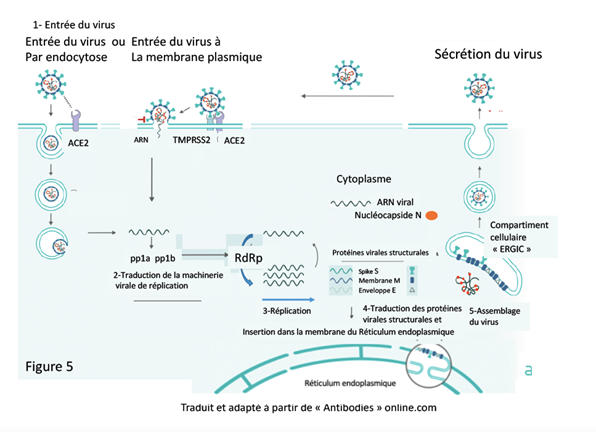
Dans une phase précoce de la traduction, deux poly-protéines précurseurs (pp1a et pp1ab) sont produites. Celles-ci possèdent une activité protéase responsable de leur auto-clivage en plusieurs protéines maturées, dites non structurales (car ne participant pas à la formation de la particule virale). Ces protéines forment le complexe réplicase-transcriptase (CRT) nécessaire à la multiplication du génome viral. Parmi elles on trouve l’ARN polymérase ARN-dépendante ou réplicase (RdRp), qui permet de faire de nouvelles copies du génome viral ARN. Au sein du CRT, de petits transcrits viraux dit subgénomiques sont aussi produits. Ils codent les protéines structurales (M, E, S et N) qui composent la particule virale. Dès qu’elles émergent des ribosomes, les protéines M, E et S sont insérées dans la membrane du réticulum endoplasmique cellulaire. La protéine N (ribonucléoprotéine) est responsable de la reconnaissance et l’empaquetage du génome viral répliqué pour former la nucléocapside. Via la protéine N la nucléocapside va aussi interagir avec la protéine M pour initier la formation de la nouvelle particule virale. Ainsi, des vésicules composées des protéines virales membranaires, et englobant la nucléocapside, émergent dans le lumen (l’intérieur) d’un compartiment dérivé du réticulum endoplasmique, appelé « ERGIC » (processus appelé bourgeonnement) (2). Au cours de cette étape la protéine S est incorporée dans la particule virale naissante. Les virus ainsi constitués sont acheminés à la surface de la cellule infectée en suivant la voie de sécrétion (appareil de Golgi, puis vésicules sécrétoires) puis libérés dans le milieu extracellulaire par « exocytose », prêts à infecter d’autres cellules.
Le virus détourne donc à son profit tant les ribosomes et les ARNt, que tous les organelles et machineries mis en jeu dans la voie de sécrétion des protéines (acheminement du réticulum endoplasmique à la surface cellulaire ou au milieu extérieur).
Essais thérapeutiques en cours pour lutter contre le SARS-CoV-2
Environ 500 études cliniques sont en cours pour identifier des traitements efficaces de la maladie COVID-19. Une étude européenne nommée DISCOVERY est coordonnée par l’Inserm (Institut National de la Santé et de la Recherche Médicale) et concernera 3200 Patients dans plusieurs Pays Européens dont 800 en France.
L’efficacité et la sécurité de plusieurs types de molécules thérapeutiques, sélectionnées sur la base d’analyses préalables sur des cellules isolées (in vitro) et/ou des modèles animaux, seront testées par rapport à des soins standard. Parmi les options à l’étude il y a la chloroquine, un antipaludique utilisé aussi dans le traitement de maladies inflammatoires (telles que le Lupus), qui selon des études in vitro empêcherait la glycosylation (addition de sucres) du récepteur ACE2 utilisé lors de l’entrée virale, conduisant à l’inhibition de cette étape. Ce composé agirait surtout en inhibant aussi l’acidification des compartiments endosomaux, nécessaire lors de l’entrée du virus par endocytose. Cependant ce traitement interfère aussi avec des processus des cellules hôtes, pouvant engendrer des effets secondaires graves pour des traitements de longue durée. Parmi les médicaments testés figurent aussi le Remdesivir (utilisé contre le virus Ebola), qui inhibe la RdRp virale, arrêtant la synthèse de nouveaux génomes viraux, ou la combinaison Lopinavir et Ritonavir (utilisés dans le traitement de l’infection par le VIH), qui inhibent l’activité protéase nécessaire pour la coupure de la polyprotéine virale pp1a). D’autres essais cliniques incluent l’effet du Camostat, un inhibiteur de la protéine cellulaire TMPRS22 (4) ainsi que des molécules, telles que l’interféron beta, agissant directement sur la réponse immunitaire au virus.
Sources
- Conférence du Pr P. Sansonetti au Collège de France https://www.college-de-france.fr/site/actualites/Covid-19ChroniqueEmergenceAnnoncee.htm
- Fehr and Perlman Meth Mol Biol 1282 1 (2015)
- https://presse.inserm.fr/lancement-dun-essai-clinique-europeen-contre-le-covid-19/38737/
- Hoffmann, M et al, 2020 Cell 181 271 (2020)
https://www.thelancet.com/journals/landig/article/PIIS2589-7500(20)30086-8/fulltext
